Artykuł pt. Hybridization and embryological patterns underpinning reproductive barriers in Dactylorhiza (Orchidaceae)
Autorami pracy są badacze naszego Wydziału - dr Aleksandra Naczk (Katedra Taksonomii Roślin i Ochrony Przyrody), dr Joanna Rojek (Katedra Biologii Eksperymentalnej i Biotechnologii Roślin), dr Małgorzata Kapusta (Laboratorium Bioobrazowania), dr hab. Marcin Górniak (Katedra Genetyki Ewolucyjnej i Biosystematyki), a także dr hab. Bożena Kolano, prof. UŚ (Uniwersytet Śląski), dr Katarzyna Meyza (Uniwersytet Kazimierza Wielkiego w Bydgoszczy) oraz dr Joanna Znaniecka (MWB UG i GUMed).
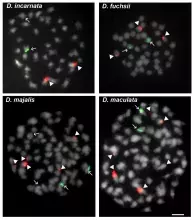
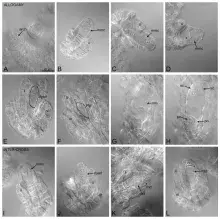
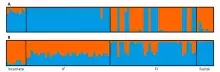
W niniejszym badaniu (https://www.nature.com/articles/s41598-025-24871-2) zbadano mechanizmy izolacji reprodukcyjnej w obrębie kompleksu Dactylorhiza incarnata/maculata przy użyciu metod cytologicznych, embriologicznych i molekularnych. Fluorescencyjna hybrydyzacja in situ ujawniła specyficzne dla gatunku wzorce rDNA. Badania embriologiczne w kontrolowanych warunkach zapylania wykazały zmienny rozwój gametofitu i żywotność nasion. Krzyżówki międzygatunkowe między diploidalnymi gatunkami rodzicielskimi dały asymetryczne hybrydy, przy czym krzyżówki między D. fuchsii × D. incarnata wykazały znacznie wyższy wskaźnik powodzenia. Analiza cytologiczna ujawniła nieprawidłowości mejozy, w tym zaburzenia segregacji chromosomów podczas megasporogenezy u D. incarnata i D. maculata, co sugeruje, że mechanizmy cytogenetyczne mogą zmniejszać płodność i żywotność nasion. Analiza markerów mikrosatelitarnych hybryd F1 ujawniła wpływ gatunku macierzyńskiego na genotypy hybryd. Wszystkie te wyniki sugerują, że bariery postzygotyczne dominują nad mechanizmami prezygotycznymi w tym złożonym kompleksie, podważając dotychczasowe założenia dotyczące kierunku powstawania allopoliploidów w tej grupie. Nasze wyniki rzucają nowe światło na mechanizmy reprodukcyjne leżące u podstaw powtarzających się zjawisk hybrydyzacji i poliploidyzacji, które ukształtowały różnorodność w rodzaju Dactylorhiza.
Fig.1. Rozmieszczenie loci 35S i 5S rDNA na chromosomach metafazy mitotycznej badanych gatunków Dactylorhiza. Hybrydyzację fluorescencyjną in situ przeprowadzono przy użyciu sond 25S rDNA (fluorescencja czerwona) i 5S rDNA (fluorescencja zielona).
Fig.2. Rozwój zalążków po allogamii i krzyżowaniu międzygatunkowym. Allogamia (A–H); krzyżowanie międzygatunkowe (I–L).
Fig.3. Analiza STRUCTURE z wykorzystaniem diploidalnych gatunków rodzicielskich Dactylorhiza i ich hybryd F1.